在做生物信息的过程中,经常需要进行各种文件格式。每一种生物软件都有固定的文件格式要求。因此,需要非常每一种数据的文件格式,从某种意义上来说,生物信息分析的过程就是进行各种文件格式的转换过程。例如当前很多分析都可以概括为从fastq到bam,从bam到vcf的过程。
fasta文件格式
FASTA文件主要用于存储生物的序列文件,例如基因组,基因的核酸序列以及氨基酸等,是最常见的生物序列格式,一般以扩展名fa,fasta,fna等。fasta文件中,第一行是由大于号”>”开头的任意文字说明,用于序列标记,为了保证后续分析软件能够区分每条序列,单个序列的标识必须是唯一的,序列ID部分可以包含注释信息。从第二行开始为序列本身,只允许使用既定的核苷酸或氨基酸编码符号。序列部分可以在一行,也可以分成多行。
>gi|556503834|ref|NC_000913.3|:190-255 Escherichia coli str. K-12 substr. MG1655, complete genome
ATGAAACGCATTAGCACCACCATTACCACCACCATCACCATTACCACAGGTAACGGTGCGGGCTGA
>gi|556503834|ref|NC_000913.3|:337-2799 Escherichia coli str. K-12 substr. MG1655, complete genome
娜娜项目网每日更新创业和副业项目
网址:nanaxm.cn 点击前往娜娜项目网
站 长 微 信: nanadh666
ATGCGAGTGTTGAAGTTCGGCGGTACATCAGTGGCAAATGCAGAACGTTTTCTGCGTGTTGCCGATATTC
TGGAAAGCAATGCCAGGCAGGGGCAGGTGGCCACCGTCCTCTCTGCCCCCGCCAAAATCACCAACCACCT
GGTGGCGATGATTGAAAAAACCATTAGCGGCCAGGATGCTTTACCCAATATCAGCGATGCCGAACGTATT
TTTGCCGAACTTTTGACGGGACTCGCCGCCGCCCAGCCGGGGTTCCCGCTGGCGCAATTGAAAACTTTCG
TCGATCAGGAATTTGCCCAAATAAAACATGTCCTGCATGGCATTAGTTTGTTGGGGCAGTGCCCGGATAG
fastq文件格式
fastq文件格式是用来存储测序文件的,它是含有quality的fasta文件。
@DJB775P1:248:D0MDGACXX:7:1202:12362:49613
TGCTTACTCTGCGTTGATACCACTGCTTAGATCGGAAGAGCACACGTCTGAA
JJJJJIIJJJJJJHIHHHGHFFFFFFCEEEEEDBD?DDDDDDBDDDABDDCA
@DJB775P1:248:D0MDGACXX:7:1202:12782:49716
CTCTGCGTTGATACCACTGCTTACTCTGCGTTGATACCACTGCTTAGATCGG
IIIIIIIIIIIIIIIHHHHHHFFFFFFEECCCCBCECCCCCCCCCCCCCCCC
质量值体系

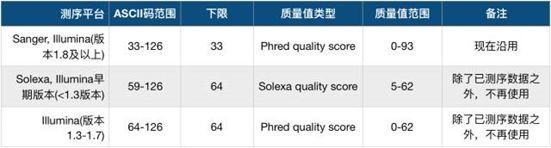
从表中可以看到下限有33和64两个值,我们把加33的的质量值体系称之为Phred33,加64的称之为Phred64(Solexa的除外,它叫Selexa64)。不过,现在一般都是使用Phred33这个体系,而且33也恰好是ASCII的第一个可见字符('!')
sam格式介绍
sam文件主要用来存储短序列比对的结尾,即将测序数据定位到基因组上的表示形式。
第一列:是reads ID
第二列:是flag标记的总和
第三列:比对到参考序列上的染色体号。
第四列:为在参考序列上的位置
第五列:比对的质量值,MAPQ
第六列:代表比对结果的CIGAR字符串
第七列:mate比对到的染色体号,若是没有mate,则是*
第八列:比对到参考序列上的第一个碱基位置
第九列:Template的长度文档格式,
第十列:为read的序列
第十一列:为ASCII码格式的序列质量;
VCF文件格式介绍
VCF是Variant Call Format的简称,是一种定义的专门用于存储基因序列突变信息的文本格式。在生物信息分析中会大量用到VCF格式。例如基因组中的单碱基突变,SNP, 插入/缺失INDEL, 拷贝数变异CNV,和结构变异SV等文档格式,都是利用VCF格式来存储的。将其存储为二进制格式就是BCF。
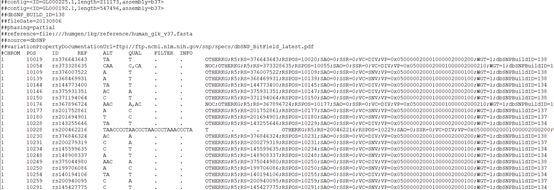
1.CHROM [chromosome]: 染色体名称,
2.POS [position]: 参考基因组突变碱基位置,如果是INDEL,位置是INDEL的第一个碱基位置。
3.ID [identifier]: 突变的名称,
4.REF [reference base(s)]:参考染色体的碱基
5.ALT [alternate base(s)]: 与参考序列比较,发生突变的碱基,
6.QUAL [quality]: Phred标准下的质量值
7.FILTER [filter status]:使用其它的方法进行过滤后得到的过滤结果
8.INFO
文件格式介绍:#format1
娜娜项目网每日更新创业和副业项目
网址:nanaxm.cn 点击前往娜娜项目网
站 长 微 信: nanadh666

